Capítulo 3 Estructura de los datos
3.1 Formato ancho y largo
Una vez que hemos recogido nuestros datos, debemos proceder con un análisis descriptivo para saber qué modelo debemos usar con el fin de contestar a las preguntas científicas planteadas en el estudio. La organización y visualización de los datos en estudios longitudinales no es tan sencilla como en cualquier otro tipo de diseño ya que los datos se pueden organizar de formas distintas. Básicamente, podemos:
Datos a nivel de individuo, en el que cada individuo tiene un registro y múltiples variables contienen los datos de cada ocasión de medición (datos en formato ancho - wide format en inglés).
Datos a nivel de tiempo, en el que cada persona tiene varios registros, uno para cada ocasión de medición (datos en formato largo - long format en inglés).
Las funciones que tenemos en R tanto para visualizar como modelizar datos longitudinales puede requerir el tener los datos en cualquiera de los dos formatos. El formato largo normalmente se necesita para agrupar variables como por ejemplo si queremos visualizar nuestra información mediante gráficos de barras apilados. En R existen muchas funciones para pasar de formato ancho a largo y viceversa. Recientemente se han creado las fuciones dcast() y melt() en la librería reshape2 que facilitan enormemente estas tareas. La siguiente figura muestra un ejemplo de cómo utilizar estas funciones:
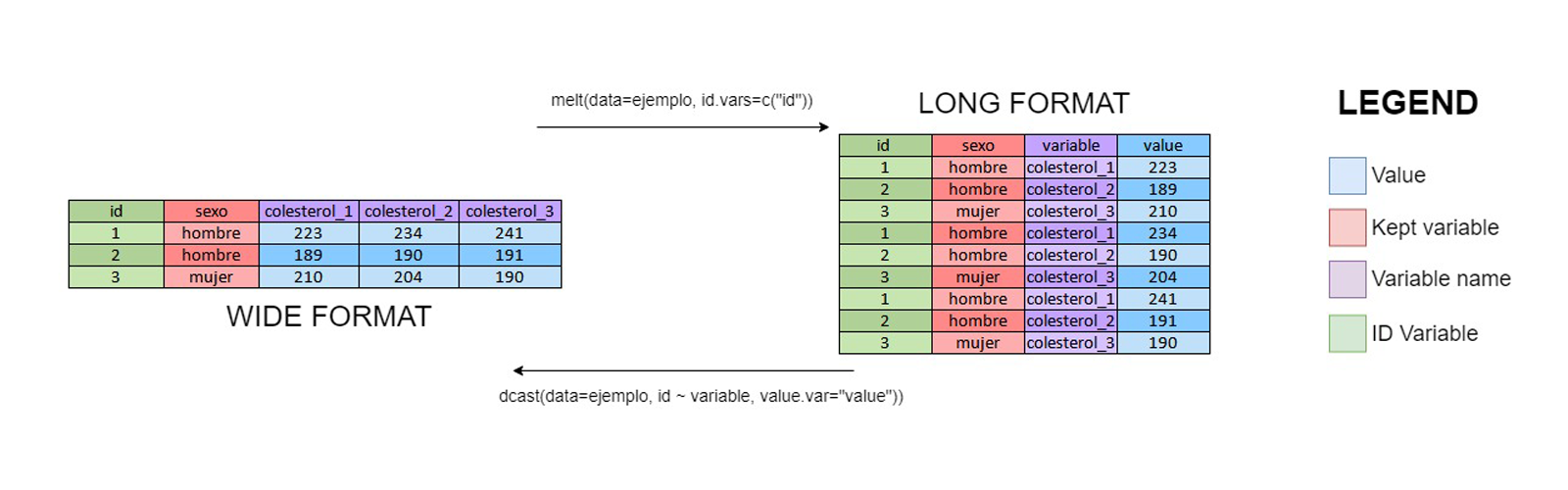
Figura 3.1: Datos en formato ancho y largo y cómo pasar de un formato a otro usando funciones de la librería reshape2
Veamos cómo realizarlo con R. Empecemos por cargar los datos que hemos visto en el ejemplo anterior
datos <- read.delim("datos/ejemplo.txt")
datos id sexo colesterol_1 colesterol_2 colesterol_3
1 1 hombre 223 234 241
2 2 hombre 189 190 191
3 3 mujer 210 204 190Vemos que están en formato ancho. Podemos pasarlos a formato largo utilizando la función melt() que tiene los siguientes argumentos:
dataes el objeto (data.frame o tibble) que vamos a convertir de ancho a largo,id.varsson las variables en la tabla que vamos a dejar sin cambiar de dimensión. En nuestro ejemplo sería la variable “sexo”, aunque pueden ser más en tablas más complejas. Puede usarse un vector de nombres (tipo character) o de números enteros que correspondan al número de columna.measure.varsson las variables en las que se encuentran las mediciones. Puede ser un vector de nombres o de números enteros indicando los índices de las columnas. En nuestro caso son las columnas 3 a 5.variable.namees el nombre que va a adoptar la columna en la que queden nuestras variables, es decir, en nuestro caso sería el tiempo. Por defecto usa “variable”.value.namees el nombre que va a adoptar la columna en la que queden los valores, que en nuestro caso sería el colestero. Por defecto usa “value”.variable.factores una valor lógico (TRUE o FALSE) para indicar si queremos que la columna de variable quede convertida a factor (opción por defecto), o quede simplemente como caracter.
Veamos cómo aplicamos esto a nuestro ejemplo
library(reshape2)
datos_largo <- melt(datos, measure.vars=3:5,
variable.name = "tiempo",
value.name = "colesterol")
datos_largo id sexo tiempo colesterol
1 1 hombre colesterol_1 223
2 2 hombre colesterol_1 189
3 3 mujer colesterol_1 210
4 1 hombre colesterol_2 234
5 2 hombre colesterol_2 190
6 3 mujer colesterol_2 204
7 1 hombre colesterol_3 241
8 2 hombre colesterol_3 191
9 3 mujer colesterol_3 190Notemos que nuestra variable tiempo no es numérica indicando el momento donde se toma la medida de colesterol. Debería de ser una variable numérica 1, 2, 3. Podemos solucionar esto eliminando “colesterol_” de la variable simplemente ejecutando:
library(tidyverse)
datos_largo <- mutate(datos_largo,
tiempo = str_remove(tiempo, "colesterol_") %>%
as.numeric())
datos_largo id sexo tiempo colesterol
1 1 hombre 1 223
2 2 hombre 1 189
3 3 mujer 1 210
4 1 hombre 2 234
5 2 hombre 2 190
6 3 mujer 2 204
7 1 hombre 3 241
8 2 hombre 3 191
9 3 mujer 3 190Podemos ordenar nuestros datos por individuo y tiempo de la siguiente manera
datos_largo <- arrange(datos_largo, id, tiempo)
datos_largo id sexo tiempo colesterol
1 1 hombre 1 223
2 1 hombre 2 234
3 1 hombre 3 241
4 2 hombre 1 189
5 2 hombre 2 190
6 2 hombre 3 191
7 3 mujer 1 210
8 3 mujer 2 204
9 3 mujer 3 190Veamos ahora cómo pasar de formato largo a ancho. Para esta tarea usamos la función dcast(). Esta función tiene una notación un poco diferente, pues usa fórmulas para determinar qué variables poner en cada lugar. Tiene los siguientes argumentos:
dataes la tabla que vamos a convertir,formulaes la forma en que vamos a distribuir las columnas. En general la fórmula es de forma x ~ y. Se puede usar una regla nemotécnica que consiste en: filas ~ columnas.dropdeberían los valores faltantes ser eliminados o mantenidos?. Por defecto es TRUE y no se ponen.value.vares el nombre (o número) de la columna en la que están los valores. Generalmentedcast()adivina bien este valor, pero es bueno usarlo para asegurarnos de lo que estamos haciendo y evitar que salga un mensaje de advertencia.
En nuestro caso ejecutaríamos:
datos_ancho <- dcast(datos_largo, id ~ tiempo,
value.var = "colesterol")
datos_ancho id 1 2 3
1 1 223 234 241
2 2 189 190 191
3 3 210 204 190Si queremos mantener el resto de covariables debemos ejecutar:
datos_ancho <- dcast(datos_largo, id + sexo ~ tiempo,
value.var = "colesterol")
datos_ancho id sexo 1 2 3
1 1 hombre 223 234 241
2 2 hombre 189 190 191
3 3 mujer 210 204 190que es justo el conjunto de datos inicial del que partíamos excepto por el nombre de las variables (que ahora se llaman 1, 2, 3). Para poder poner el nombre original, basta con ejecutar:
datos_ancho <- dcast(datos_largo, id + sexo ~ paste0("colesterol_", tiempo),
value.var = "colesterol")
datos_ancho id sexo colesterol_1 colesterol_2 colesterol_3
1 1 hombre 223 234 241
2 2 hombre 189 190 191
3 3 mujer 210 204 190Veamos ahora ejemplos más reales donde tenemos más de una variable repetida a lo largo del tiempo, datos faltantes u datos recogidos en distintos tiempos.
3.2 Formato ancho
Como hemos comentado anteriormente, lo normal es recoger los datos en formato ancho (u horizontal) data su simplicidad.
| ind | sexo | edad | coltot_1 | coltot_2 | coltot_3 | bmi_1 | bmi_2 | bmi_3 |
|---|---|---|---|---|---|---|---|---|
| 1 | 1 | 44 | 213 | 220 | 199 | 33.3 | 30.9 | 28.3 |
| 2 | 0 | 45 | 196 | 238 | 218 | 31.1 | 29.1 | 30.1 |
| 3 | 1 | 55 | 195 | 218 | 216 | 28.5 | 30.0 | 27.9 |
| 4 | 0 | 51 | 201 | 194 | 201 | 32.5 | 31.4 | 24.5 |
| 5 | 1 | 46 | 234 | 185 | 189 | 30.1 | 31.9 | 27.7 |
| 6 | 0 | 51 | 213 | 183 | 214 | 30.4 | 28.8 | 28.3 |
La ventaja de esta estrategia es que tenemos una fila para cada individuo, como estamos acostumbrados.
Sin embargo, existen varios inconvenientes:
Si tenemos un missing en alguna medida hay que eliminar a todo el individuo
Debemos suponer que todas las medidas se han realizado en los mismos momentos para todos los individuos, y esto puede no ser cierto.
Las diferentes medidas de una misma variable predictora la debemos analizar como si fueran distintas variables
3.3 Formato largo
Estos mismos datos se dispondrían de la siguiente forma en formato largo (o vertical)
| ind | sexo | edad | coltot | bmi | momento |
|---|---|---|---|---|---|
| 1 | 1 | 44 | 213 | 33.3 | 1 |
| 1 | 1 | 44 | 220 | 30.9 | 2 |
| 1 | 1 | 44 | 199 | 28.3 | 3 |
| 2 | 0 | 45 | 196 | 31.1 | 1 |
| 2 | 0 | 45 | 238 | 29.1 | 2 |
| 2 | 0 | 45 | 218 | 30.1 | 3 |
| 3 | 1 | 55 | 195 | 28.5 | 1 |
| 3 | 1 | 55 | 218 | 30.0 | 2 |
| 3 | 1 | 55 | 216 | 27.9 | 3 |
| 4 | 0 | 51 | 201 | 32.5 | 1 |
| 4 | 0 | 51 | 194 | 31.4 | 2 |
| 4 | 0 | 51 | 201 | 24.5 | 3 |
| 5 | 1 | 46 | 234 | 30.1 | 1 |
| 5 | 1 | 46 | 185 | 31.9 | 2 |
| 5 | 1 | 46 | 189 | 27.7 | 3 |
| 6 | 0 | 51 | 213 | 30.4 | 1 |
| 6 | 0 | 51 | 183 | 28.8 | 2 |
| 6 | 0 | 51 | 214 | 28.3 | 3 |
3.4 Valores faltantes
Cuando hay valores faltantes en una medida y los datos se disponen de forma horizontal se descartan los demás valores ya que se elimina toda la fila.
| ind | sexo | edad | coltot_1 | coltot_2 | coltot_3 | bmi_1 | bmi_2 | bmi_3 |
|---|---|---|---|---|---|---|---|---|
| 1 | 1 | 44 | 213 | 220 | 199 | 33.3 | 30.9 | 28.3 |
| 2 | 0 | 45 | 196 | NA | 218 | 31.1 | 29.1 | 30.1 |
| 3 | 1 | 55 | 195 | 218 | 216 | 28.5 | 30.0 | 27.9 |
| 4 | 0 | 51 | 201 | 194 | 201 | 32.5 | 31.4 | 24.5 |
| 5 | 1 | 46 | 234 | 185 | NA | 30.1 | 31.9 | 27.7 |
| 6 | 0 | 51 | 213 | 183 | 214 | 30.4 | 28.8 | 28.3 |
En cambio, en la disposición vertical sólo se pierden los valores de los tiempos en cuestión y no todas las medidas del individuo.
| ind | sexo | edad | coltot | bmi | momento |
|---|---|---|---|---|---|
| 1 | 1 | 44 | 213 | 33.3 | 1 |
| 1 | 1 | 44 | 220 | 30.9 | 2 |
| 1 | 1 | 44 | 199 | 28.3 | 3 |
| 2 | 0 | 45 | 196 | 31.1 | 1 |
| 2 | 0 | 45 | NA | 29.1 | 2 |
| 2 | 0 | 45 | 218 | 30.1 | 3 |
| 3 | 1 | 55 | 195 | 28.5 | 1 |
| 3 | 1 | 55 | 218 | 30.0 | 2 |
| 3 | 1 | 55 | 216 | 27.9 | 3 |
| 4 | 0 | 51 | 201 | 32.5 | 1 |
| 4 | 0 | 51 | 194 | 31.4 | 2 |
| 4 | 0 | 51 | 201 | 24.5 | 3 |
| 5 | 1 | 46 | 234 | 30.1 | 1 |
| 5 | 1 | 46 | 185 | 31.9 | 2 |
| 5 | 1 | 46 | NA | 27.7 | 3 |
| 6 | 0 | 51 | 213 | 30.4 | 1 |
| 6 | 0 | 51 | 183 | 28.8 | 2 |
| 6 | 0 | 51 | 214 | 28.3 | 3 |
3.5 Tiempos de medidas diferentes
Al disponer los datos de forma vertical se puede especificar en qué momento se ha recogido cada medida. Para ello simplemente se indica en la variable tiempo. Por ejemplo si se trata de los días que han pasado desde el momento inicial del experimento.
| ind | sexo | edad | coltot | bmi | momento | dias |
|---|---|---|---|---|---|---|
| 1 | 1 | 44 | 213 | 33.3 | 1 | 1 |
| 1 | 1 | 44 | 220 | 30.9 | 2 | 4 |
| 1 | 1 | 44 | 199 | 28.3 | 3 | 5 |
| 2 | 0 | 45 | 196 | 31.1 | 1 | 3 |
| 2 | 0 | 45 | 238 | 29.1 | 2 | 7 |
| 2 | 0 | 45 | 218 | 30.1 | 3 | 10 |
| 3 | 1 | 55 | 195 | 28.5 | 1 | 2 |
| 3 | 1 | 55 | 218 | 30.0 | 2 | 7 |
| 3 | 1 | 55 | 216 | 27.9 | 3 | 8 |
| 4 | 0 | 51 | 201 | 32.5 | 1 | 2 |
| 4 | 0 | 51 | 194 | 31.4 | 2 | 8 |
| 4 | 0 | 51 | 201 | 24.5 | 3 | 9 |
| 5 | 1 | 46 | 234 | 30.1 | 1 | 1 |
| 5 | 1 | 46 | 185 | 31.9 | 2 | 9 |
| 5 | 1 | 46 | 189 | 27.7 | 3 | 10 |
| 6 | 0 | 51 | 213 | 30.4 | 1 | 3 |
| 6 | 0 | 51 | 183 | 28.8 | 2 | 6 |
| 6 | 0 | 51 | 214 | 28.3 | 3 | 10 |
O incluso podemos tener mas medidas para unos individuos que para otros. Como sería el caso que tuviéramos algún missing en alguna medida. Como en este ejemplo, para el individuo 2 y el 5 tenemos sólo 2 medidas, mientras que para el resto tenemos 3.
| ind | sexo | edad | coltot | bmi | momento | dias |
|---|---|---|---|---|---|---|
| 1 | 1 | 44 | 213 | 33.3 | 1 | 1 |
| 1 | 1 | 44 | 220 | 30.9 | 2 | 4 |
| 1 | 1 | 44 | 199 | 28.3 | 3 | 5 |
| 2 | 0 | 45 | 196 | 31.1 | 1 | 3 |
| 2 | 0 | 45 | 218 | 30.1 | 3 | 10 |
| 3 | 1 | 55 | 195 | 28.5 | 1 | 2 |
| 3 | 1 | 55 | 218 | 30.0 | 2 | 7 |
| 3 | 1 | 55 | 216 | 27.9 | 3 | 8 |
| 4 | 0 | 51 | 201 | 32.5 | 1 | 2 |
| 4 | 0 | 51 | 194 | 31.4 | 2 | 8 |
| 4 | 0 | 51 | 201 | 24.5 | 3 | 9 |
| 5 | 1 | 46 | 234 | 30.1 | 1 | 1 |
| 5 | 1 | 46 | 185 | 31.9 | 2 | 9 |
| 6 | 0 | 51 | 213 | 30.4 | 1 | 3 |
| 6 | 0 | 51 | 183 | 28.8 | 2 | 6 |
| 6 | 0 | 51 | 214 | 28.3 | 3 | 10 |
3.6 Transformación
3.6.1 Vertical a horizontal y viceversa
En esta sección aprovecharemos para ver otras instrucciones útiles en R para pasar de la disposición vertical de los datos a la horizontal y viceversa (aunque yo recomiendo usar melt() y dcast(). Para ello usaremos los datos del ejemplo anterior que están guardados en formato .csv de la siguiente manera:
tablahorizontal <- read.csv2("datos/tablahorizontal.csv")
tablahorizontal ind sexo edad coltot_1 coltot_2 coltot_3 bmi_1 bmi_2 bmi_3
1 1 1 44 213 220 199 33.3 30.9 28.3
2 2 0 45 196 238 218 31.1 29.1 30.1
3 3 1 55 195 218 216 28.5 30.0 27.9
4 4 0 51 201 194 201 32.5 31.4 24.5
5 5 1 46 234 185 189 30.1 31.9 27.7
6 6 0 51 213 183 214 30.4 28.8 28.3Como tenemos la base de datos en horizontal (una fila por individuo) y la queremos pasar a vertical (un registro por fila y varias filas por individuo) podemos usar:
tablong <- reshape(data=tablahorizontal,
direction="long",
varying=list(c("coltot_1","coltot_2","coltot_3"),
c("bmi_1","bmi_2","bmi_3")),
times=1:3,
timevar="momento",
idvar="ind",
v.names=c("coltot","bmi"))
tablong ind sexo edad momento coltot bmi
1.1 1 1 44 1 213 33.3
2.1 2 0 45 1 196 31.1
3.1 3 1 55 1 195 28.5
4.1 4 0 51 1 201 32.5
5.1 5 1 46 1 234 30.1
6.1 6 0 51 1 213 30.4
1.2 1 1 44 2 220 30.9
2.2 2 0 45 2 238 29.1
3.2 3 1 55 2 218 30.0
4.2 4 0 51 2 194 31.4
5.2 5 1 46 2 185 31.9
6.2 6 0 51 2 183 28.8
1.3 1 1 44 3 199 28.3
2.3 2 0 45 3 218 30.1
3.3 3 1 55 3 216 27.9
4.3 4 0 51 3 201 24.5
5.3 5 1 46 3 189 27.7
6.3 6 0 51 3 214 28.3Ordeno la tabla por id y dentro de cada id por tiempo
tablong <- arrange(tablong, ind, momento)
tablong ind sexo edad momento coltot bmi
1.1 1 1 44 1 213 33.3
1.2 1 1 44 2 220 30.9
1.3 1 1 44 3 199 28.3
2.1 2 0 45 1 196 31.1
2.2 2 0 45 2 238 29.1
2.3 2 0 45 3 218 30.1
3.1 3 1 55 1 195 28.5
3.2 3 1 55 2 218 30.0
3.3 3 1 55 3 216 27.9
4.1 4 0 51 1 201 32.5
4.2 4 0 51 2 194 31.4
4.3 4 0 51 3 201 24.5
5.1 5 1 46 1 234 30.1
5.2 5 1 46 2 185 31.9
5.3 5 1 46 3 189 27.7
6.1 6 0 51 1 213 30.4
6.2 6 0 51 2 183 28.8
6.3 6 0 51 3 214 28.3Y si queremos pasar del formato largo al horizontal
tablavertical <- read.csv2("datos/tablavertical.csv")
tablavertical ind sexo edad coltot bmi momento dias
1 1 1 44 213 33.3 1 1
2 1 1 44 220 30.9 2 4
3 1 1 44 199 28.3 3 5
4 2 0 45 196 31.1 1 3
5 2 0 45 238 29.1 2 7
6 2 0 45 218 30.1 3 10
7 3 1 55 195 28.5 1 2
8 3 1 55 218 30.0 2 7
9 3 1 55 216 27.9 3 8
10 4 0 51 201 32.5 1 2
11 4 0 51 194 31.4 2 8
12 4 0 51 201 24.5 3 9
13 5 1 46 234 30.1 1 1
14 5 1 46 185 31.9 2 9
15 5 1 46 189 27.7 3 10
16 6 0 51 213 30.4 1 3
17 6 0 51 183 28.8 2 6
18 6 0 51 214 28.3 3 10tabwide <- reshape(data=tablavertical,
direction="wide",
v.names=c("coltot","bmi"),
times=1:3,
timevar="momento",
idvar="ind")
tabwide ind sexo edad dias coltot.1 bmi.1 coltot.2 bmi.2 coltot.3 bmi.3
1 1 1 44 1 213 33.3 220 30.9 199 28.3
4 2 0 45 3 196 31.1 238 29.1 218 30.1
7 3 1 55 2 195 28.5 218 30.0 216 27.9
10 4 0 51 2 201 32.5 194 31.4 201 24.5
13 5 1 46 1 234 30.1 185 31.9 189 27.7
16 6 0 51 3 213 30.4 183 28.8 214 28.3¿Y si tenemos algun individuo con menos medidas? Por ejemplo, tenemos la tabla en formato vertical y para el individuo id=1 tenemos dos medidas en lugar de 3 (quitamos la tercera medida)
tablaverticalmiss <- tablavertical[-3,]
tablaverticalmiss ind sexo edad coltot bmi momento dias
1 1 1 44 213 33.3 1 1
2 1 1 44 220 30.9 2 4
4 2 0 45 196 31.1 1 3
5 2 0 45 238 29.1 2 7
6 2 0 45 218 30.1 3 10
7 3 1 55 195 28.5 1 2
8 3 1 55 218 30.0 2 7
9 3 1 55 216 27.9 3 8
10 4 0 51 201 32.5 1 2
11 4 0 51 194 31.4 2 8
12 4 0 51 201 24.5 3 9
13 5 1 46 234 30.1 1 1
14 5 1 46 185 31.9 2 9
15 5 1 46 189 27.7 3 10
16 6 0 51 213 30.4 1 3
17 6 0 51 183 28.8 2 6
18 6 0 51 214 28.3 3 10tabwidemiss <- reshape(data=tablaverticalmiss,
direction="wide",
v.names=c("coltot","bmi"),
times=1:3,
timevar="momento",
idvar="ind")
tabwidemiss ind sexo edad dias coltot.1 bmi.1 coltot.2 bmi.2 coltot.3 bmi.3
1 1 1 44 1 213 33.3 220 30.9 NA NA
4 2 0 45 3 196 31.1 238 29.1 218 30.1
7 3 1 55 2 195 28.5 218 30.0 216 27.9
10 4 0 51 2 201 32.5 194 31.4 201 24.5
13 5 1 46 1 234 30.1 185 31.9 189 27.7
16 6 0 51 3 213 30.4 183 28.8 214 28.33.6.2 Colapsar
Si tenemos los datos en vertical y queremos colapsar o resumir los distintos datos de cada individuo en un único valor, como por ejemplo la media.
library(dplyr)
library(magrittr)
group_by(tablavertical, ind) %>%
summarise_at(vars(coltot, bmi), list(media = mean))# A tibble: 6 x 3
ind coltot_media bmi_media
<int> <dbl> <dbl>
1 1 211. 30.8
2 2 217. 30.1
3 3 210. 28.8
4 4 199. 29.5
5 5 203. 29.9
6 6 203. 29.2